Vi beskriver här översiktligt hur processerna går till enligt AquaBiotas eDNA-system.
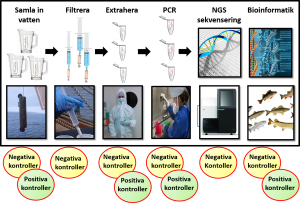
Flödesschema som visar hur eDNA provtagningen går till från insamling till analys av flera arter i taget (metabarcoding).
1. Insamling av prover
Innan vattnet samlas in utförs planering för att optimera provpunkternas antal och placering utifrån syftet med studien. Vattnets egenskaper mäts under provtagning; temperatur, pH och många andra miljöfaktorer registreras, eftersom dessa påverkar hur länge eDNA finns kvar i vattnet. Denna tid kan variera från några dagar till några veckor. Felaktig studiedesign, exempelvis gällande var och hur proverna samlas in, kan leda till resultat som inte är användbara. Därför är designen av studien mycket viktig för eDNA-projekt.
Vattnet samlas in i steriliserade provtagningskärl av speciellt tränad personal. Det är mycket viktigt att använda handskar och andra rutiner så att man inte kontaminerar proverna. Negativa och positiva kontroller läggs till vid alla kritiska steg för att säkerställa att slutresultaten är korrekta. Vatten filtreras på plats genom speciella filter, mellan 1-5 liter beroende på filter och partikelkoncentration. Filtren fixeras sedan i en konserveringsvätska på plats som bevarar DNA-partiklarnas struktur. Proven transporteras sedan till laboratorier för fortsatta analys.
2. Utvinning av eDNA
AquaBiota har tillsammans med MoRe Research i Örnsköldsvik inrättat ett sterilt laboratorium som enbart används för att utvinna eDNA. DNA utvinns där med hjälp av samma principer som man använder i ett kriminaltekniskt laboratorium. Alla inblandade bär munskydd och heltäckande dräkter under arbetets gång. Detta är viktigt eftersom alla kontamineringsrisker måste undvikas. Färdigt utvunnet DNA har en volym av 1/10 000 L och detta räcker till många analyser. DNA sparas i särskilda utrymmen tills de processas vidare.
3. Enarts- och flerarts- analyser
Beroende på frågeställningen kan vi med hjälp av molekylära analyser detektera endera en art i taget eller flera arter på en gång. Större vattensalamander är ett exempel på undersökning vi utfört för en enskild art. Undersökningar som t.ex. skall ta reda på vilka fiskarter eller groddjur som finns i ett givet vattendrag använder flerartsanalyser vilket betyder att man ta reda på alla de arter av fisk eller groddjur som finns i ett prov på en gång.
3a. Enartsanalyser – Barcoding
Då man vill ta reda på om en viss art, t.ex. större vattensalamander, finns i en damm eller sjö utförs enartsanalyser (barcoding). Principen bygger på att varje levande art utsöndrar arvsmassa eller DNA. Vissa delar av en individs DNA är helt unikt för just den individen, medan andra delar av DNA delas med just den individens art, och sedan finns det DNA som ser likadant ut hos alla organismer i en grupp. För enartsanalyser väljer man ut en kort bit av DNA som är unik för den valda arten. Denna artspecifika bit DNA tillverkas syntetiskt och kallas primer. Dessa primers, en droppe eDNA, ett enzym och salter blandas i ett provrör. Provröret placeras i en maskin som gör massor av DNA kopior för just den arten. Resultatet, dvs. DNA-sekvensen som primern kopierar kallas barcode (streckkod), och kan liknas vid en streckkod som används för att betala för varor i affärer. Maskinerna man använder för att kopiera DNA utnyttjar samma processer som sker i alla levande celler och kallas för qPCR (quantitative Polymerase Chain Reaction) och droplet PCR. Medan provet körs mäts hur mycket DNA som kopieras, och om arten man söker finns i DNA-provet ger detta ett kvantitativt utslag. Om arten inte finns i provet ges ingen signal. Antalet kopior indikerar hur mycket det finns av en art. Provsvaren kommer upp genast efter körningen.
3b. Flerartsanalyser – Metabarcoding genom Next Generation Sequencing (Massive Parallel Sequencing)
Metabarcoding är en ypperlig metod då man vill få reda på alla arter som finns i ett vatten med hjälp av eDNA. Genom en enda analys kan man få fram tusentals arter. Principen för metabarcoding är densamma som för barcoding för en art. Primerna för metabarcoding tillverkas så att de kan kopiera upp en bit DNA som är likadan för alla arter inom en grupp, tex alla fiskar, men även ett avsnitt som varierar mellan arter och visar en unik kod. Blandningen av DNA, primer, salt och enzym används för att kopiera DNA genom en PCR-maskin. För att avläsa resultaten körs proverna igenom en så kallad NGS (Next Generation Sequencing) analys, där man får fram de artspecifika DNA sekvenserna.
4. Flerartsanalyser- bioinformatik
Då resultaten är klara har man enorma mängder data som motsvarar miljontals olika kopior av DNA. Ett enda prov ger mer än 1 terabyte data. För att få ordning på denna data och för att veta vilka arter man har i ett prov använder man olika analytiska metoder som sorterar upp data. Enorma datafiler analyseras med hjälp av program som skiljer de olika DNA-koderna åt. Denna process kallas bioinformatik. Då bioinformatiken är klar och sekvenserna har sorterats skapas en lista av olika DNA-identiteter. Dessa matchas mot olika databaser och varje unik bit av DNA får en identitet. Slutresultatet är en datatabell över identifierade arter och antalet DNA-matchningar för varje art. Man kan då säga vilka arter som finns i en sjö och den relativa förekomsten av just den arten. Metabarcoding av ett vattenprov ger möjligheter att analysera både tidigare kända och okända arter som finns i ett vattendrag på kort tid.
5. Flerartsanlyser- ekologisk tolkning av resultat
Specialister på AquaBiota går igenom artlistorna och analyserar de olika arternas förekomst och den ekologiska betydelsen. Okända, sällsynta och även ovälkomna främmande arter kan påträffas i proverna. Vanligtvis hittar vi arter som traditionell provtagningsmetodik missar och dessutom betydligt fler arter. Exempelvis visade sig stensimpa vara vanlig i Norrbotten och vi har identifierat den invasiva arten Silverkarp i ett vattendrag i Östergötland.
Granskning av resultatet inkludera även en genetisk bedömning, där vi tittar på likhetsgraden inom en gen för att se att artlistan är korrekt. Om detta steget skippas kan arter försvinna från slutresultatet, tillföljd av felaktiga sammanslagningar av arter som har en höglikhetsgrad inom den analyserade genen. Viket exempelvis förekommer bland plattfiskar som analyseras inom den ofta använda 12S regionen. Därutöver använder vi också geografisk bedömning och analyser mellan primers för att säkerställa att sekvenser korrekt artsätts när taxonomiska konflikter uppstår i analysen.
Eftersom eDNA-provtagningen i fält går betydligt snabbare än traditionella metoder kan stora geografiska områden undersökas på kort tid. Undersökningar av eDNA som upprepas med jämna tidsintervaller är mycket användbara för att verifiera miljöförändringar över tid, eller om arter försvinner eller återetableras vid olika åtgärder så som dammbyggen eller restaureringsåtgärder. Resultaten relaterar väl till sammanslaget data från mångåriga intensiva provfisken. eDNA ger en betydligt mer komplett bild av artförekomster än traditionella inventeringar.